 Problem 6: Molekylærbiologi har unnlatt å gi et stort "Livets tre"
Problem 6: Molekylærbiologi har unnlatt å gi et stort "Livets tre"
Oversatt herfra.
Når fossiler ikke viste at dyr utviklet seg fra en felles forfedre, vendte evolusjonære forskere seg til en annen type bevis - DNA-sekvensdata - for å demonstrere livets tre. I 1960-tallet, rundt den tiden den genetiske koden ble forstått, forutse biokjemikene Émile Zuckerkandl og Linus Pauling at hvis DNA-sekvenser kunne brukes til å produsere evolusjonære trær - trær som matchet dem basert på morfologiske eller anatomiske egenskaper - ville dette gi "det beste tilgjengelig enkelt-bevis på realiteten av makro-evolusjon." (99) Således begynte en flere tiår lang innsats for å sekvensere gener av mange organismer og konstruere "molekylære" baserte evolusjonære ("fylogenetiske") trær. Det endelige målet har vært å bygge et stort "livets tre", som viser hvordan alle levende organismer er relatert gjennom universell felles avstamning.
Hovedforutsetningen
Den grunnleggende logikken bak å bygge molekylærtrær er relativt enkel. Først velger forskerne et gen, eller et sett av gener, funnet på tvers av flere organismer. Deretter analyseres disse genene for å bestemme deres nukleotidsekvenser, slik at gen-sekvensene til forskjellige organismer da kan sammenlignes. Endelig er et evolusjonært tre konstruert basert på prinsippet om at jo mer nukleotidsekvensen ligner, jo nærmere relatert er artene. En artikkel i tidsskriftet Biologisk teori sier det slik:
Molekylær systematikk er (i stor grad) basert på antagelsen, først tydelig formulert av Zuckerkandl og Pauling (1962), graden av overordnet likhet gjenspeiler grad av relaterthet. (100)
Denne antakelsen er i hovedsak en artikulasjon av et hovedtrekk i teorien - ideen om universell felles avstamning. Ikke desto mindre er det viktig å innse at det bare er en antagelse å påstå at genetiske 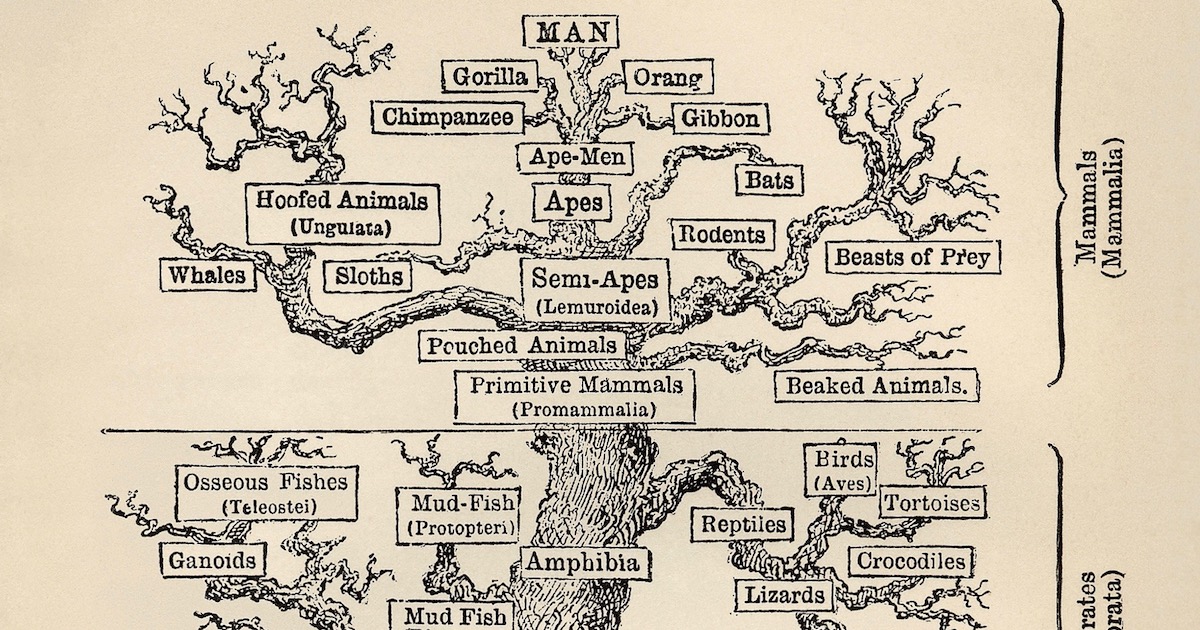 likheter mellom forskjellige arter nødvendigvis skyldes felles forfedre.
likheter mellom forskjellige arter nødvendigvis skyldes felles forfedre.
Strengt fungerende innenfor et darwinistisk paradigme, flyter disse forutsetningene naturlig. Som forutnevnte 'Biological Theory' artikkel forklarer, stammer hovedforutsetningen som ligger til grunn for molekylære trær "fra tolkning av molekylær likhet (eller ulikhet) mellom taxa, i sammenheng med en darwinistisk modell av kontinuerlig og gradvis endring." (101) Så teorien antas å være sann for å bygge et tre. Men også: Hvis darwinistisk evolusjon er sann, bør konstruksjon av trær ved hjelp av forskjellige sekvenser avsløre et rimelig konsistent mønster over forskjellige gener eller sekvenser.
Dette gjør det enda betyderligere at innsatsen for å bygge et stort "livets tre" ved hjelp av DNA eller andre biologiske sekvensdata, ikke har vært i samsvar med forventningene. Det grunnleggende problemet er at ett gen gir én versjon av livets tre, mens et annet gen gir en svært forskjellig og motstridende versjon av treet. For eksempel, som vi vil diskutere nærmere nedenfor, plasserer stabdard pattedyr-treet mennesker nærmere knyttet til gnagere enn til elefanter. Men studier av en bestemt type DNA kalt mikro-RNA-gener har antydet det motsatte - at menneskene var nærmere elefanter enn gnagere. Slike konflikter mellom gen-baserte trær er svært vanlige.
De genetiske dataene maler derfor ikke et konsekvent bilde av felles forfedre, og viser at antagelsene om trebygging ofte mislykkes. Dette fører til forsvarlige spørsmål hvorvidt universell felles avstamning er riktig.
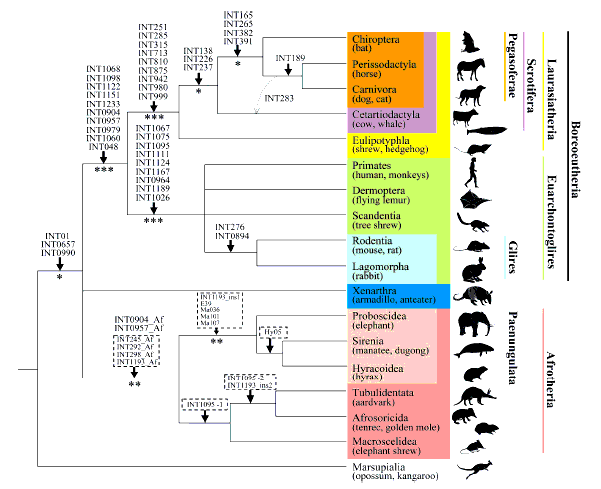 Konflikter i basen av livets tre
Konflikter i basen av livets tre
Problemer oppstod først da molekylærbiologer sekvenserte gener fra de tre grunnleggende domenene i livet - bakterier, arkea og eukaryoter - men de genene tillot ikke at disse grunnleggende gruppene av liv ble løst i et tre-lignende mønster. I 2009 publiserte tidsskriftet New Scientist en coverstory med tittelen "Hvorfor Darwin tok feil om livets tre", som forklarte disse problemene:
-Problemene begynte tidlig på 1990-tallet da det ble mulig å sekvensere faktiske bakterielle og arkeale gener fremfor bare RNA. Alle ventet at disse DNA-sekvensene skulle bekrefte RNA-treet, og noen ganger gjorde de det, men avgjørende, noen ganger gjorde de det ikke. RNA, for eksempel, kan foreslå at arten A var nærmere relatert til art B enn arten C, men et tre laget av DNA ville foreslå det motsatte. (102)
Denne typen data førte til at biokjemikeren W. Ford Doolittle forklarte at "Molekylære fylogenister vil ha mislyktes med å finne det 'sanne treet', ikke fordi deres metoder er utilstrekkelige eller fordi de har valgt feil gener, men fordi livets historie ikke ordentlig kan bli representert som et tre." (103) New Scientist sa det slik: "I lang tid, var den hellige gralen å bygge et livets tre ... Men i dag ligger prosjektet i biter, revet i stykker fra angrep av negative bevis." (104)
Mange evolusjonister svarer noen ganger at disse problemene bare oppstår når man studerer mikroorganismer som bakterier - organismer som kan bytte gener gjennom en prosess som kalles 'horisontal genoverføring', og derved fordunkler signalet om evolusjonære relasjoner. Men denne innvendingen er ikke helt sann, siden livets tre utfordres selv blant høyere organismer der slike genbytter ikke er utbredt. Carl Woese, en pioner for evolusjonær molekylær systematikk, forklarer:
'Fylogenetiske inkongruenser kan sees overalt i det universelle treet, fra roten til de store forgreningene i og blant de forskjellige taxaene til istandsettelsen av selve de primære grupperingene.' (105)
På samme måte bemerker New Scientist- artikkelen at 'forskning tyder på at utviklingen av dyr og planter ikke akkurat er tre-lik heller.' (106) Artikkelen forklarer hva som skjedde da mikrobiologen Michael Syvanen forsøkte å lage et tre som viser evolusjonære relasjoner ved hjelp av 2000 gener fra en variert gruppe dyr:
-Han feilet. Problemet var at forskjellige gener fortalte motstridende evolusjonære historier. ... genene sendte blandede signaler. ... Omtrent 50 prosent av genene har én evolusjonær historie og 50 prosent en annen. (107)
Dataene var så vanskelige å veksle inn i et tre at Syvanen beklaget: "Vi har nettopp tilintetgjort livets tre." (108) Mange andre artikler i teknisk litteratur gjenkjenner lignende problemer.
Konflikter mellom høyere grener
Et 2009-artikkel i Trends in Ecology and Evolution viser at "En stor utfordring for å inkorporere så store mengder data i slutninger om arts-trær er at motstridende slektshistorier ofte eksisterer i forskjellige gener gjennom genomet." (109) På samme måte studerte en artikkel i Genome Research DNA-sekvensene i ulike dyregrupper og fant at 'forskjellige proteiner genererer forskjellige fylogenetiske treer'. (110) En juni-artikkel fra 2012 i Nature rapporterte at korte tråder av RNA kalt mikro-RNAs 'river i stykker tradisjonelle ideer om dyre-treet.' Dartmouth biolog Kevin Peterson som studerer microRNAs klaget: "Jeg har sett på tusenvis av mikroRNA gener, og Jeg kan ikke finne et enkelt eksempel som ville støtte det tradisjonelle treet." Ifølge artikkelen ga microRNAs "et radikalt annet diagram for pattedyr: en som justerer mennesker nærmere til elefanter enn med gnagere. "Peterson sa det utelukkende:" MicroRNAs er helt entydige ... de gir et helt annet tre fra hva alle andre ønsker. " (111)
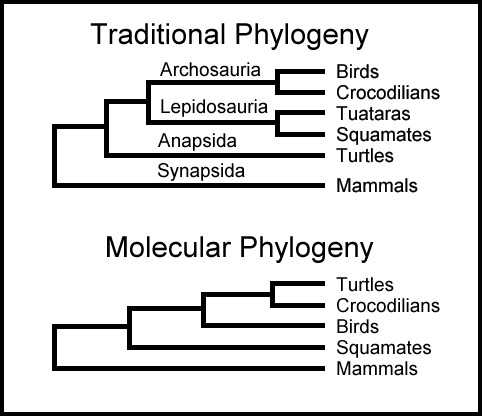 Konflikter mellom molekyler og morfologi
Konflikter mellom molekyler og morfologi
Ikke alle fylogenetiske trær er konstruert ved å sammenligne molekyler som DNA fra forskjellige arter. Mange trær er basert på å sammenligne form, struktur og kroppsplan for forskjellige organismer - også kalt 'morfologi'. Men konflikter mellom molekylbaserte trær og morfologi-baserte trær er også vanlige. En artikkel fra 2012 som studerer flaggermus-relasjoner gjorde dette klart, og erklærer: "Inkongruens mellom fylogenier avledet fra morfologiske versus molekylære analyser, og mellom trær basert på forskjellige undergrupper av molekylære sekvenser er blitt gjennomgripende, da datasettene har ekspandert raskt i både karaktertrekk og arter." (112) Dette er neppe den eneste studien som støter på konflikter mellom DNA-baserte trær og trær basert på anatomiske eller morfologiske egenskaper. Lærebøker hevder ofte at felles avstamning støttes ved hjelp av eksemplet på et dyretre basert på enzymet cytokrom c som matcher det tradisjonelle evolusjonære treet basert på morfologi. (113) Imidlertid nevner lærebøker sjelden at treet basert på et annet enzym, cytokrom b , er i sterk konflikt med det vanlige evolusjonære treet. Som en artikkel i Trends in Ecology and Evolution observerte:
Det mitokondrielle cytokrom b-genet indikerte. . . en absurd fylogeni av pattedyr, uavhengig av metoden for trekonstruksjon. Katter og hvaler falt i primat-gruppen, gruppert med simians (apekatter og store aper) og strepsirhines (lemurer, bush-babyer og marekatter) til utelukkelse av tarsierer. Cytokrom b er sannsynligvis det mest sekvenserte genet i virveldyr, noe som gjør dette overraskende resultatet enda mer forvirrende. (114)
Påfallende nok, konkluderte en annen artikkel i Trends in Ecology and Evolution med at "riket av konkurrerende morfologiske, samt molekylære forslag [av] de fremherskende fylogeniene i pattedyr rekkene ville redusere pattedyrtreet til en uløselig bush, det eneste konsekvente [evolusjonære forhold] er sannsynligvis gruppering av elefanter og sjøkyr." (115) På grunn av slike konflikter, rapporterte en hoved-vurderingsartikkel i Nature at "ulikheter mellom molekylære og morfologiske trær" fører til 'evolusjonskrig' fordi "evolusjonære trær konstruert ved å studere biologiske molekyler ofte ikke ligner de som er bygget opp fra morfologi." (116)
Endelig forsøkte en studie publisert i Science i 2005 å bruke gener til å rekonstruere forholdene til dyre-fylaene, men konkluderte med at "til tross for mengden data og bredden av taxa analysert, forble forholdet mellom de fleste [animalske phyla] ikke-løst. "Det følgende år publiserte de samme forfatterne en vitenskapelig artikkel med tittelen" Bushes in the Tree of Life ", som ga slående konklusjoner. Forfatterne erkjenner at "en stor del av enkeltgener produserer fylogenier av dårlig kvalitet," og observerte at den ene studien "utelot 35% av enkeltgener fra sin datamatrise, fordi de genene produserte fylogenier i strid med konvensjonell visdom." Papiret antyder at "Visse kritiske deler av livets tre kan være vanskelig å finne ut av, uavhengig av mengden av konvensjonelle data som er tilgjengelige." Papiret hevder likevel at "den gjentatte oppdagelsen av vedvarende uløste clader, burde tvinge frem en reevaluering av flere utbredt antagelser om molekylær systematikk. " (117)
Dessverre er en antagelse som disse evolusjonære biologene ikke er villige til å revurdere, antagelsen om at universell felles avstamning er riktig. De appellerer til et myriade av ad hoc argumenter - horisontal genoverføring, lang grenattraksjon, rask utvikling, forskjellige utviklingshastigheter, koaleserende teori, ufullstendig prøvetaking, feilaktig metodologi og konvergent evolusjon - for å forklare ubeleilige data som ikke passer til ettertraktede tre-like mønster. Som en 2012-artikkel uttalte, er "fylogenetisk konflikt vanlig, og ofte normen i stedet for unntaket." (118) På slutten av dagen har drømmen om at DNA-sekvensdata skulle passe inn i et pent og pyntelig livsens tre mislyktes, og med det en nøkkelforutsigelse for neo-darwinistisk teori.
 Referanser:
Referanser:
[99.] Zuckerkandl and Pauling, "Evolutionary Divergence and Convergence in Proteins," 101.
[100.] Jeffrey H. Schwartz, Bruno Maresca, "Do Molecular Clocks Run at All? A Critique of Molecular Systematics," Biological Theory, 1(4):357-371, (2006). [101.] Ibid.
[102.] Graham Lawton, "Why Darwin was wrong about the tree of life," New Scientist (January 21, 2009).
[103.] W. Ford Doolittle, "Phylogenetic Classification and the Universal Tree," Science, 284:2124-2128 (June 25, 1999).
[104.] Partly quoting Eric Bapteste, in Lawton, "Why Darwin was wrong about the tree of life" (internal quotations omitted).
[105.] Carl Woese "The Universal Ancestor," Proceedings of the National Academy of Sciences USA, 95:6854-9859 (June, 1998) (emphasis added).
[106.] Graham Lawton, "Why Darwin was wrong about the tree of life," New Scientist (January 21, 2009).
[107.] Partly quoting Michael Syvanen, in Lawton, "Why Darwin was wrong about the tree of life" (internal quotations omitted).
[108.] Michael Syvanen, quoted in Lawton, "Why Darwin was wrong about the tree of life."
[109.] James H. Degnan and Noah A. Rosenberg, "Gene tree discordance, phylogenetic inference and the multispecies coalescent," Trends in Ecology and Evolution, 24 (2009): 332-340.
[110.] Arcady R. Mushegian, James R. Garey, Jason Martin and Leo X. Liu, "Large-Scale Taxonomic Profiling of Eukaryotic Model Organisms: A Comparison of Orthologous Proteins Encoded by the Human, Fly, Nematode, and Yeast Genomes," Genome Research, 8 (1998): 590-598.
[111.] Elie Dolgin, "Rewriting Evolution," Nature, 486: 460-462 (June 28, 2012).
[112.] Liliana M. Dávalos, Andrea L. Cirranello, Jonathan H. Geisler, and Nancy B. Simmons, "Understanding phylogenetic incongruence: lessons from phyllostomid bats," Biological Reviews of the Cambridge Philosophical Society, 87:991-1024 (2012).
[113.] For example, see BSCS Biology: A Molecular Approach (Glencoe/McGraw Hill, 2006), 227; Sylvia S. Mader, Jeffrey A. Isaacson, Kimberly G. Lyle-Ippolito, Andrew T. Storfer, Inquiry Into Life, 13th ed. (McGraw Hill, 2011), 550.
[114.] See Michael S. Y. Lee, "Molecular Phylogenies Become Functional," Trends in Ecology and Evolution, 14: 177 (1999).
[115.]W. W. De Jong, "Molecules remodel the mammalian tree," Trends in Ecology and Evolution, 13(7), pp. 270-274 (July 7, 1998).
[116.] Trisha Gura, "Bones, Molecules or Both?," Nature, 406 (July 20, 2000): 230-233
[117.] Antonis Rokas & Sean B. Carroll, "Bushes in the Tree of Life," PLoS Biology, 4(11): 1899-1904 (Nov., 2006) (internal citations and figures omitted).
[118.] Liliana M. Dávalos, Andrea L. Cirranello, Jonathan H. Geisler, and Nancy B. Simmons, "Understanding phylogenetic incongruence: lessons from phyllostomid bats," Biological Reviews of the Cambridge Philosophical Society, 87:991-1024 (2012).
Oversettelse og bilder ved Asbjørn E. Lund
 Problem 6: Molekylærbiologi har unnlatt å gi et stort "Livets tre"
Problem 6: Molekylærbiologi har unnlatt å gi et stort "Livets tre" Problem 6: Molekylærbiologi har unnlatt å gi et stort "Livets tre"
Problem 6: Molekylærbiologi har unnlatt å gi et stort "Livets tre" likheter mellom forskjellige arter nødvendigvis skyldes felles forfedre.
likheter mellom forskjellige arter nødvendigvis skyldes felles forfedre.  Konflikter i basen av livets tre
Konflikter i basen av livets tre Konflikter mellom molekyler og morfologi
Konflikter mellom molekyler og morfologi Referanser:
Referanser: